- La PCR és un mètode utilitzat per amplificar l'ADN a partir d'una petita quantitat de plantilla d'ADN.La RT-PCR utilitza la transcripció inversa per produir una plantilla d'ADN a partir d'una font d'ARN que després es pot amplificar.
- La PCR i la RT-PCR solen ser reaccions finals, mentre que la qPCR i la RT-qPCR utilitzen la cinètica de la velocitat de síntesi del producte durant la reacció de PCR per quantificar la quantitat de plantilla present.
- Els mètodes més nous, com la PCR digital, proporcionen una quantificació absoluta de la plantilla inicial d'ADN, mentre que mètodes com la PCR isotèrmica redueixen la necessitat d'equips cars per proporcionar resultats fiables.
La reacció en cadena de la polimerasa (PCR) és una tècnica de biologia molecular relativament senzilla i àmpliament utilitzada per amplificar i detectar seqüències d'ADN i ARN.En comparació amb els mètodes tradicionals de clonació i amplificació d'ADN, que sovint poden trigar dies, la PCR només requereix unes poques hores.La PCR és altament sensible i requereix una plantilla mínima per a la detecció i l'amplificació de seqüències específiques.Els mètodes bàsics de PCR han avançat encara més des de la detecció simple d'ADN i ARN.A continuació, us oferim una visió general dels diferents mètodes de PCR i dels reactius que oferim a Enzo Life Sciences per a les vostres necessitats de recerca.El nostre objectiu és ajudar els científics a accedir ràpidament als reactius de PCR per utilitzar-los en el seu proper projecte de recerca!
PCR
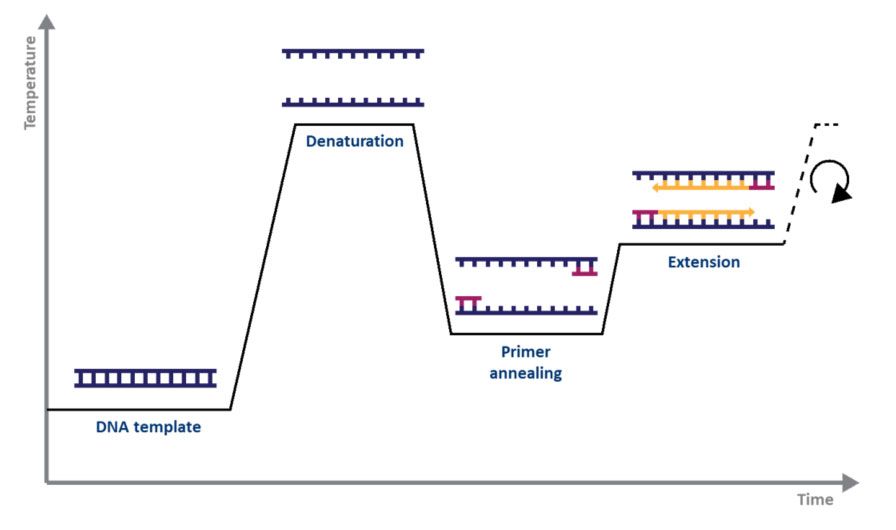
Per a la PCR estàndard, tot el que necessiteu és una ADN polimerasa, magnesi, nucleòtids, cebadors, la plantilla d'ADN que s'ha d'amplificar i un termociclador.El mecanisme de PCR és tan senzill com el seu propòsit: 1) l'ADN de doble cadena (dsDNA) es desnaturalitza per calor, 2) els cebadors s'alineen a les cadenes simples d'ADN i 3) els cebadors s'estenen per l'ADN polimerasa, donant com a resultat dues còpies de l'ADN. cadena d'ADN original.El procés de desnaturalització, recuit i allargament durant una sèrie de temperatures i temps es coneix com un cicle d'amplificació (Fig. 1).
| Figura 1.Representació esquemàtica d'un cicle d'amplificació per PCR. |
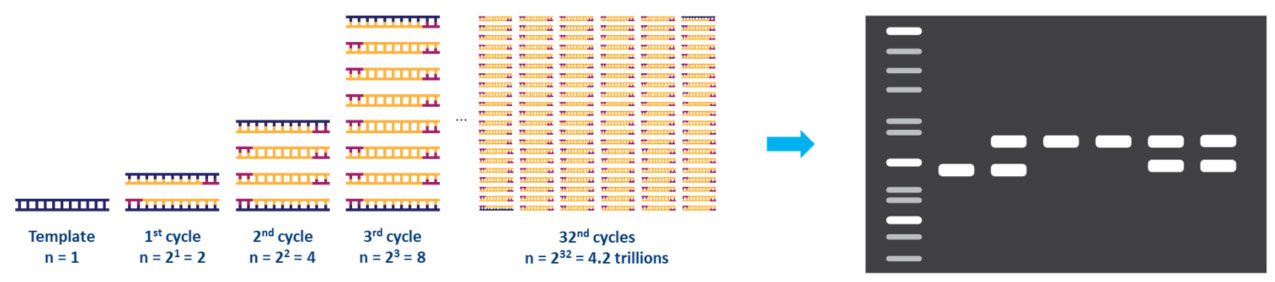
Cada pas del cicle s'ha d'optimitzar per a la plantilla i el conjunt d'imprimacions utilitzats.Aquest cicle es repeteix aproximadament 20-40 vegades, i després es pot analitzar el producte amplificat, normalment amb gel d'agarosa (Fig. 2).
| Figura 2.Amplificació d'una plantilla d'ADN per PCR i anàlisi per electroforesi en gel d'agarosa. |
Com que la PCR és un mètode molt sensible i es requereixen volums molt petits per a reaccions individuals, es recomana la preparació d'una mescla mestra per a diverses reaccions.La mescla mestra s'ha de barrejar bé i després dividir-se pel nombre de reaccions, assegurant-se que cada reacció contindrà la mateixa quantitat d'enzim, dNTPs i cebadors.Molts proveïdors, com Enzo Life Sciences, també ofereixen barreges de PCR que ja contenen tot, excepte primers i la plantilla d'ADN.
Les regions riques en guanina/citosina (riques en GC) representen un repte en les tècniques estàndard de PCR.Les seqüències riques en GC són més estables que les seqüències amb menor contingut en GC.A més, les seqüències riques en GC tendeixen a formar estructures secundàries, com ara bucles de forquilla.Com a resultat, les cadenes dobles riques en GC són difícils de separar completament durant la fase de desnaturalització.En conseqüència, l'ADN polimerasa no pot sintetitzar la nova cadena sense obstacles.Una temperatura de desnaturalització més alta pot millorar-ho, i els ajustaments cap a una temperatura de recuit més alta i un temps de recuit més curt poden evitar la unió inespecífica dels primers rics en GC.Els reactius addicionals poden millorar l'amplificació de seqüències riques en GC.El DMSO, el glicerol i la betaïna ajuden a interrompre les estructures secundàries que són causades per les interaccions GC i, per tant, faciliten la separació de les cadenes dobles.
PCR d'inici en calent
L'amplificació inespecífica és un problema que pot ocórrer durant la PCR.La majoria de les ADN polimerases que s'utilitzen per a la PCR funcionen millor a temperatures d'entre 68 °C i 72 °C.L'enzim, però, també pot ser actiu a temperatures més baixes, encara que en un grau inferior.A temperatures molt inferiors a la temperatura de recuit, els primers poden unir-se de manera inespecífica i conduir a una amplificació inespecífica, fins i tot si la reacció es configura en gel.Això es pot evitar mitjançant l'ús d'inhibidors de la polimerasa que es dissocien de l'ADN polimerasa només un cop s'arriba a una determinada temperatura, d'aquí el terme PCR d'inici en calent.L'inhibidor pot ser un anticòs que s'uneix a la polimerasa i es desnaturalitza a la temperatura de desnaturalització inicial (normalment 95 °C).
Polimerasa d'alta fidelitat
Tot i que les ADN polimerases s'amplifiquen amb força precisió a la seqüència de plantilla original, es poden produir errors en la concordança de nucleòtids.Els desajustaments en aplicacions com la clonació poden donar lloc a transcripcions truncades i proteïnes traduïdes incorrectament o inactives aigües avall.Per evitar aquests desajustaments, s'han identificat polimerases amb activitat de "correcció de proves" i s'han incorporat al flux de treball.La primera polimerasa de correcció de proves, Pfu, es va identificar l'any 1991 a Pyrococcus furiosus.Aquest enzim Pfu té una activitat exonucleasa de 3' a 5'.A mesura que l'ADN s'amplifica, l'exonucleasa elimina els nucleòtids no coincidents a l'extrem 3' de la cadena.Aleshores es substitueix el nucleòtid correcte i continua la síntesi d'ADN.La identificació de seqüències de nucleòtids incorrectes es basa en l'afinitat d'unió pel nucleòsid trifosfat correcte amb l'enzim, on una unió ineficient retarda la síntesi i permet la substitució correcta.L'activitat de correcció de proves de la polimerasa Pfu provoca menys errors en la seqüència final en comparació amb la DNA polimerasa Taq.En els darrers anys, s'han identificat altres enzims de correcció de proves i s'han fet modificacions de l'enzim Pfu original per reduir encara més la taxa d'error durant l'amplificació de l'ADN.
RT-PCR
La PCR de transcripció inversa, o RT-PCR, permet l'ús de l'ARN com a plantilla.Un pas addicional permet la detecció i l'amplificació de l'ARN.L'ARN es transcriu inversament a ADN complementari (ADNc), mitjançant la transcriptasa inversa.La qualitat i la puresa de la plantilla d'ARN són essencials per a l'èxit de la RT-PCR.El primer pas de la RT-PCR és la síntesi d'un híbrid ADN/ARN.La transcriptasa inversa també té una funció RNasa H, que degrada la porció d'ARN de l'híbrid.Aleshores, la molècula d'ADN monocatenari es completa amb l'activitat de l'ADN polimerasa depenent de l'ADN de la transcriptasa inversa en ADNc.L'eficiència de la reacció de la primera cadena pot afectar el procés d'amplificació.A partir d'aquí, s'utilitza el procediment estàndard de PCR per amplificar l'ADNc.La possibilitat de revertir l'ARN en ADNc mitjançant RT-PCR té molts avantatges i s'utilitza principalment per a l'anàlisi de l'expressió gènica.L'ARN és monocatenari i molt inestable, cosa que fa que sigui difícil treballar-hi.Normalment serveix com a primer pas en qPCR, que quantifica les transcripcions d'ARN en una mostra biològica.
qPCR i RT-qPCR
La PCR quantitativa (qPCR) s'utilitza per detectar, caracteritzar i quantificar àcids nucleics per a nombroses aplicacions.En RT-qPCR, les transcripcions d'ARN sovint es quantifiquen mitjançant la transcripció inversa primer a cDNA, tal com es descriu anteriorment, i després es realitza la qPCR.Com en la PCR estàndard, l'ADN s'amplifica mitjançant tres passos que es repeteixen: desnaturalització, recuit i allargament.Tanmateix, en qPCR, l'etiquetatge fluorescent permet la recollida de dades a mesura que avança la PCR.Aquesta tècnica té molts beneficis a causa de la varietat de mètodes i químiques disponibles.
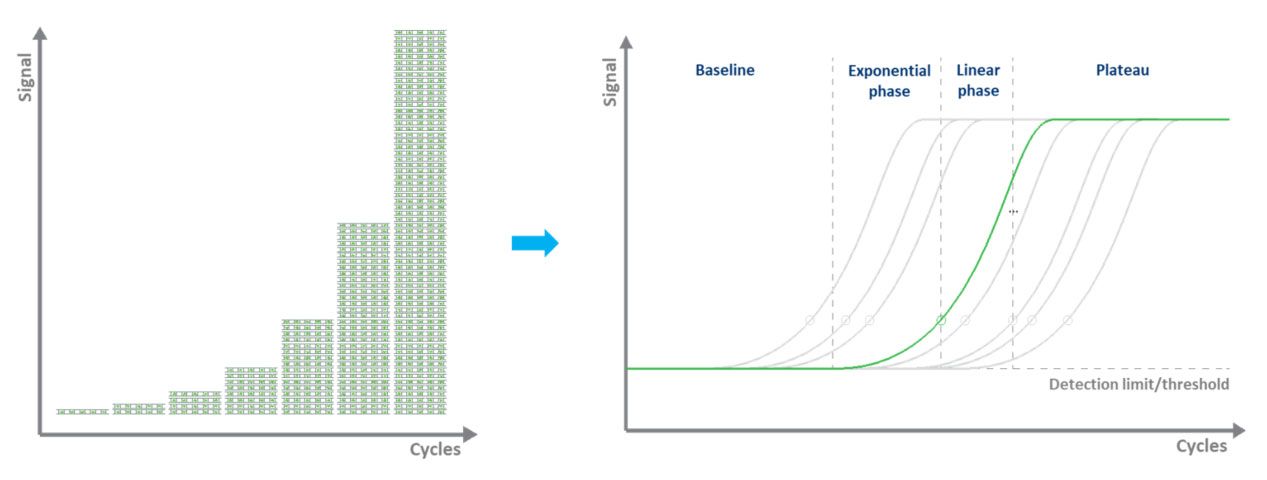
En qPCR basat en colorants (normalment verd), l'etiquetatge fluorescent permet la quantificació de les molècules d'ADN amplificades mitjançant l'ús d'un colorant d'unió dsDNA.Durant cada cicle es mesura la fluorescència.El senyal de fluorescència augmenta proporcionalment a la quantitat d'ADN replicat.Per tant, l'ADN es quantifica en "temps real" (Fig. 3).Els desavantatges de la qPCR basada en colorants són que només es pot examinar un objectiu alhora i que el colorant s'uneix a qualsevol ADN-ds present a la mostra.
| Figura 3.Amplificació d'una plantilla d'ADN mitjançant qPCR i mesura del senyal de fluorescència en temps real. |
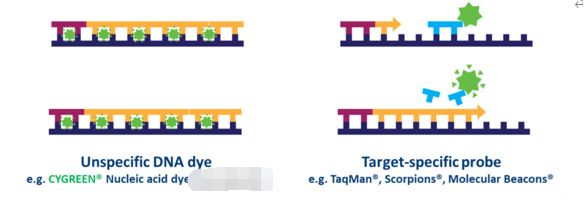
En la qPCR basada en sondes, es poden detectar moltes dianes simultàniament a cada mostra, però això requereix l'optimització i el disseny d'una o més sondes específiques de la diana utilitzades a més dels cebadors.Hi ha diversos tipus de dissenys de sondes disponibles, però el tipus més comú és una sonda d'hidròlisi, que incorpora un fluoròfor i un extintor.La transferència d'energia de ressonància de fluorescència (FRET) impedeix l'emissió del fluoròfor a través del quencher mentre la sonda està intacta.Tanmateix, durant la reacció de PCR, la sonda s'hidrolitza durant l'extensió de l'encebador i l'amplificació de la seqüència específica a la qual està unida.L'escissió de la sonda separa el fluoròfor del quencher i dóna lloc a un augment de la fluorescència depenent de l'amplificació (Fig. 4).Així, el senyal de fluorescència d'una reacció qPCR basada en sonda és proporcional a la quantitat de la seqüència objectiu de la sonda present a la mostra.Com que la qPCR basada en sondes és més específica que la qPCR basada en colorants, sovint és la tecnologia que s'utilitza en els assajos de diagnòstic basats en qPCR.
| Figura 4.Diferències entre qPCR basat en colorants i basat en sondes. |
Amplificació isotèrmica
Les tècniques de PCR esmentades anteriorment requereixen equips de termociclatge cars per augmentar i baixar amb precisió les temperatures de la cambra per als passos de desnaturalització, recuit i extensió.S'han desenvolupat una sèrie de tècniques que no necessiten dispositius tan precisos i que es poden realitzar en un simple bany maria o fins i tot dins de les cèl·lules d'interès.Aquestes tècniques s'anomenen col·lectivament amplificació isotèrmica i funcionen basades en l'amplificació exponencial, lineal o en cascada.
El tipus més conegut d'amplificació isotèrmica és l'amplificació isotèrmica mediada per bucle, o LAMP.LAMP utilitza l'amplificació exponencial a 65⁰C per amplificar l'ADN o l'ARN plantilla.Quan es realitza LAMP, de quatre a sis cebadors complementaris a les regions de l'ADN objectiu s'utilitzen amb una ADN polimerasa per sintetitzar ADN nou.Dos d'aquests cebadors tenen seqüències complementàries que reconeixen les seqüències en els altres cebadors i les uneixen, permetent que es formi una estructura de "bucle" a l'ADN recentment sintetitzat que ajudi a l'ancoratge dels cebadors en les rondes d'amplificació posteriors.LAMP es pot visualitzar mitjançant diversos mètodes, com ara fluorescència, electroforesi en gel d'agarosa o colorimetria.La facilitat per visualitzar i detectar la presència o absència de producte per colorimetria i la manca d'equips cars necessaris van fer de LAMP una opció adequada per a les proves de SARS-CoV-2 en zones on les proves de laboratori clínic no estaven fàcilment disponibles, o l'emmagatzematge i transport de mostres. no era factible, o en laboratoris que abans no disposaven d'equips de termociclatge per PCR.
Hora de publicació: 19-agost-2023